SYSTEMS BIOLOGY
SYSTEMS BIOLOGY.
– Il cambiamento di paradigma per le scienze della vita. Nuovi concetti e primi risultati rilevanti. Necessaria riorganizzazione delle scienze della vita. Bibliografia
La s. b., o biologia sistemica, è un nuovo campo di ricerca scientifica, fortemente interdisciplinare, rivolto alla comprensione dei processi biologici complessi (per es., ciclo cellulare, crescita, differenziamento, trasformazione neoplastica ecc.) che derivano dall’interazione dinamica di un gran numero di molecole. Essa richiede l’integrazione iterativa di dati molecolari, anche ottenuti su scala genomica, con analisi modellistico/computazionali. In questo modo si arriva a descrivere i meccanismi molecolari che sottendono ciascuna funzione biologica, e a predire come un determinato sistema biologico risponda a specifiche perturbazioni. Salute, agricoltura e processi industriali biotecnologici sono i campi per cui si attendono i risultati più rilevanti.
Il cambiamento di paradigma per le scienze della vita. – Alla fine del dicembre 1999, alla vigilia dell’ingresso nel 21° sec., la prestigiosa rivista scientifica «Nature» pubblicò un fascicolo speciale dedicato a quali potessero essere le aree disciplinari che avrebbero avuto maggiore impatto nel nuovo millennio. Un tema appare ricorrente in molti degli articoli riportati per le diverse aree del sapere scientifico: quello della complessità. Dall’astrofisica ai cambiamenti climatici, dalla sociologia alla biologia, molti campi delle scienze si trovavano alla fine del 20° sec. ad affrontare questo tema. Di particolare rilevanza un articolo (il cui primo autore, Leland Hart well, avrebbe ricevuto il premio Nobel per la medicina nel 2001) che tratta il tema della complessità biologica in modo da aprire nuove prospettive operative per affrontarla, attraverso quella che oggi viene chiamata systems biology. Prima di tutto viene presentata una riflessione sui limiti dell’approccio molecolare/riduzionistico per la comprensione di fenomeni biologici complessi, pur riconoscendone i grandi meriti nello sviluppo scientifico del 20° secolo. Si osserva infatti che una funzione biologica solo molto raramente può essere riconducibile a una specifica molecola/proteina, mentre nella gran parte dei casi essa deriva dall’interazione, organizzata e dinamica, di centinaia di molecole, le più rilevanti essendo proteine, varie molecole di RNA (RiboNucleic Acid) e piccole molecole, oltre che, ovviamente, DNA (DeoxyriboNucleic Acid). Inoltre il funzionamento di una cellula può essere disassemblato in molte diverse funzioni elementari, ciascuna delle quali, detta modulo, deriva dalle interazioni dinamiche di un certo numero di specifici componenti. I diversi moduli interagiscono tra loro secondo ‘principi costruttivi’, simili a quelli che si possono ritrovare nelle scienze sintetiche e progettuali (scienze computazionali e ingegneria): controllo a retroazione (feedback) negativa o positiva, amplificazione, sincronizzazione, robustezza, correzione dell’errore e così via. Ne deriva così che il modo migliore per strutturare le conoscenze sul ruolo di diverse molecole nel generare una funzione biologica è costruire un modello, possibilmente dinamico, capace di predire il funzionamento del sistema al variare delle condizioni di contesto. Ovviamente il modello è solo una rappresentazione simbolica della realtà, capace però di aumentare la nostra conoscenza e guidare gli interventi progettuali. Inoltre, i modelli offrono una sicura griglia di riferimento per strutturare l’enorme numero di dati sperimentali sulla composizione molecolare e sulle funzioni dei più diversi sistemi biologici nelle più varie condizioni che gli sviluppi delle scienze genomiche e post genomiche (transcrittomica, proteomica, metabolomica ecc.) stanno generando. Tanto per avere un’idea della complessità molecolare di un essere vivente basta ricordare che un organismo umano è composto da circa 50.000 miliardi di cellule che formano i diversi tessuti e organi. Ogni cellula, mediamente, contiene circa un miliardo di proteine di almeno 15.000 sequenze diverse, che largamente variano tra un tipo e l’altro di cellule. Inoltre, ogni singola proteina può essere modificata nella sua struttura da parte di un’altra proteina (per es., chinasi, fosfatasi ecc.), andando incontro a cambiamenti funzionali.
Nuovi concetti e primi risultati rilevanti. – La s. b. afferma che le funzioni biologiche complesse sono proprietà emergenti dotate di robustezza, generate da reti di molecole interagenti. Per emergenza si intende che la funzione (per es., la coniugazione di due cellule di lievito in presenza del corrispondente fattore di coniugazione) non è presente in nessuna delle molecole coinvolte, ma deriva dalla loro interazione secondo una sequenza di eventi specifica nel tempo e nello spazio. Al contrario, si può dire che la funzione di trasporto dei gas respiratori nel sangue è una funzione diretta dalla struttura dell’emoglobina. La robustezza fa riferimento alla proprietà delle funzioni biologiche di rimanere sostanzialmente inalterate al modificarsi di componenti del sistema, entro margini piuttosto ampi.
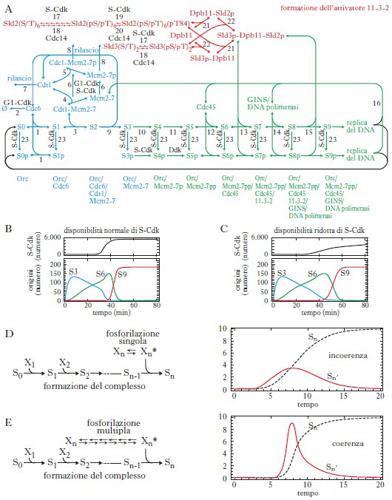
Un esempio che descrive sia emergenza sia robustezza è dato dal meccanismo di inizio della replicazione del DNA in lievito (fig. 1A). Questo processo vede un gran numero di molecole associarsi sequenzialmente a ciascuna delle circa 200 origini di replicazione presenti nel nucleo. Per una corretta e fedele duplicazione del DNA è necessario che il processo di replicazione parta contemporaneamente da ciascuna delle origini. Come viene realizzata questa sincronia?
In ognuna delle circa 200 origini di replicazione (Orc) devono venire assemblate sequenzialmente diverse proteine (per es., Cdc6, Cdt1 ecc.), ciascuna delle quali rende competente l’origine a legare la proteina successiva (fig. 1A, sequenza in basso). Anche la DNA polimerasi (che alla fine del processo catalizza la replicazione del DNA) richiede il legame precedente di una proteina, ossia il fattore 11-3-2. Questo fattore (formato da tre diverse proteine) deve essere prima fosforilato in multiple posizioni da una proteina chinasi (S-Cdk), prodotta nella tarda fase G1 che precede l’inizio della replicazione del DNA. Infatti, solo quando le componenti Sld2 e Sld3 (fig. 1A, in alto) sono completamente fosforilate divengono capaci di legarsi con il terzo partner, Dpb-11, e quindi formare il complesso attivatore 11-3-2. La simulazione del modello matematico derivato dalla rete di reazioni biochimiche della figura 1A fa emergere alcuni aspetti interessanti. La sincronia nell’accensione delle origini (fig. 1B, linea rossa S9) risulta robusta perché sostanzialmente si mantiene, anche se la disponibilità di S-Cdk si riduce considerevolmente (fig. 1B-C), mentre la formazione dei complessi intermedi S3 e S6 appare ritardata. Tale sincronia è generata come proprietà emergente della fosforilazione multipla di Sld2/Sld3, come indicato dalle simulazioni delle figure 1D e 1E, che schematizzano il processo della figura 1A.
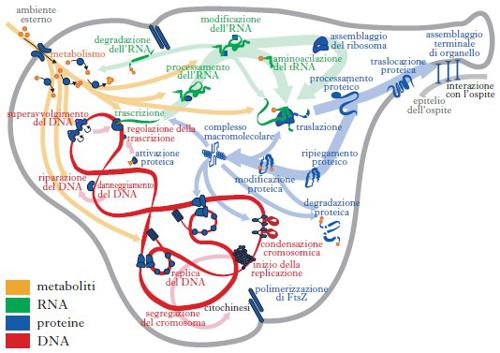
La s. b. ha raggiunto risultati molto importanti quali la costruzione del modello di un’intera cellula batterica, Mycoplasma genitalium (fig. 2): i 28 moduli del modello, ciascuno dei quali definito con diversi strumenti matematici, a seconda delle esigenze, sono stati identificati in base alle informazioni contenute in circa 900 articoli scientifici. Il modello, raffinato con 3000 simulazioni, riproduce fedelmente crescita e duplicazione cellulare. Ma la s. b. si rivolge anche ad affrontare patologie complesse, come le malattie neurodegenerative (Parkinson, Alzheimer; v. neurodegenerative, malattie), e il cancro.
Per quanto riguarda la malattia di Parkinson, è stato creato in Lussemburgo un centro di ricerche di s. b. tutto orientato verso questa patologia(http://wwwen.uni.lu/lcsb). Quando si tratta di funzioni che coinvolgono centinaia o migliaia di molecole, è opportuno procedere secondo schemi già noti da alcuni secoli alla cartografia: fare prima modelli a piccola scala, considerando moduli funzionali elementari e le loro connessioni secondo principi costruttivi, e quindi passare al livello molecolare per il modulo che, dalle precedenti analisi, appare avere un ruolo centrale nel determinare la dinamica del sistema.
Sono stati così costruiti modelli a bassa e ad alta risoluzione di eventi caratterizzanti la patologia ed è stata organizzata un’ampia comunità internazionale di ricercatori che tende ad affinare il modello e, soprattutto, a cercare di definire quali trattamenti possano rallentare il decorso della malattia.
Per il cancro si stanno percorrendo diverse strade: molti autori cercano di ricostruire, secondo la teoria dei grafi, le reti di regolazione che derivano da elaborazioni bioinformatiche di studi ‘omici’ in diverse patologie neoplastiche e di utilizzare le indicazioni derivate per sviluppare indirizzi terapeutici personalizzati. Altri studi vengono condotti, con crescente interesse, sul metabolismo di cellule tumorali confrontate con le loro controparti normali, dato che è oramai conclamato che esistono fra loro profonde differenze. Alcune società biofarmaceutiche internazionali stanno già cercando di sviluppare farmaci che abbiano come bersaglio specifico il metabolismo tumorale. Occorrerà comunque giungere a una fase di maturazione più avanzata delle ricerche per cogliere risultati veramente decisivi, capaci di curare in modo definitivo il cancro.
In conclusione, la s. b. tende a sviluppare nuovi metodi di indagine e ad acquisire nuove conoscenze sui processi biologici che sono caratterizzati da una «complessità organizzata», secondo la pionieristica definizione di Warren Weaver (1948).
Riuscire a comprendere i principi costruttivi che sottendono i mille e mille circuiti regolativi di un organismo, efficienti in quanto plasmati da miliardi di anni di evoluzione ed efficacemente rivolti al raggiungimento di uno scopo, quello di fornire un’elevata fitness evolutiva allo stesso organismo, può avere anche altri rilevanti impatti oltre a quelli precedentemente indicati. Infatti, potranno venire forniti nuovi concetti alle scienze economiche e sociali, in modo da aiutare a meglio governare i sistemi complessi costruiti dall’uomo (da quelli economico-finanziari a quelli sociopolitici) e a farli tendere, in modo più efficace, a raggiungere gli scopi positivi attesi.
Necessaria riorganizzazione delle scienze della vita. – La s. b. è interdisciplinare, ossia richiede la collaborazione di ricercatori esperti nei metodi più recenti della biologia e della chimica con altri aventi conoscenze matematiche, informatiche, ingegneristiche e fisiche, impegnati tutti insieme a ricostruire, attraverso successive iterazioni, modelli accurati delle molte funzioni complesse di cui ancora si attende di decifrare il meccanismo molecolare che le sostiene. La biologia diventa quindi big science e richiederà una profonda revisione dell’organizzazione della ricerca sia di base sia applicata, in qualche modo seguendo l’esempio tracciato dalla fisica. Anche la formazione dei ricercatori andrà modificata: si dovranno sviluppare competenze specifiche, biologiche o computazionali, ma a tutti dovrà essere data una formazione nel settore complementare, almeno sufficiente a interfacciarsi proficuamente con i colleghi. Data la complessità delle funzioni da prendere in esame, il numero dei ricercatori coinvolti potrà essere di centinaia o anche migliaia di addetti. Sorgerà quindi anche il problema di coordinare efficacemente il lavoro di questi ampi team attivi nei molti centri di s. b. che si vanno costituendo, facendoli convergere su un macro-obiettivo comune. Un modello condiviso, anche se ancora largamente incompleto, potrà servire da bussola e collante per il loro coordinamento.
Bibliografia: W. Weaver, Science and complexity, «American scientist» 1948, 36, pp. 536-44; L.H. Hartwell, J.J. Hopfield, S. Leibler et al., From molecular to modular cell biology, «Nature», 1999, 402, 6761, pp. C47-52; Systems biology. Definitions and perspectives, ed. L. Alberghina, H.V. Westerhoff, Berlin-Heidelberg 2005; H. Kitano, Towards a theory of biological robustness, «Molecular systemsbiology», 2007, 3, 137; L. Hood, S.H. Friend, Predictive, personalized, preventive, participatory (P4)cancer medicine, «Nature reviews. Clinical oncology»,2011, 8, pp. 184-87; J.R. Karr, J.C. Sanghvi, D.N. Macklin et al., A whole-cell computational model predicts phenotype from genotype, «Cell», 2012, 150, 2, pp. 389-401; G. Stephanopoulos, Synthetic biology and metabolic engineering, «ACS synthetic biology», 2012, 1, 11, pp. 514-25; T. Swierstra, N. Vermeulen, J. Braeck man et al., Rethinking the life sciences. To better serve society, biomedical research has to regain its trust and get organized to tackle larger projects, «EMBO reports», 2013, 14, 4, pp. 310-14; J. Nielsen, M. Fussenegger, J. Keasling et al., Engineering synergy in biotechnology, «Nature chemical biology», 2014, 10, 5, pp. 319-22; L. Yang, T. Moss, L.S. Mangala et al., Metabolic shifts toward glutamine regulate tumor growth, invasion and bioenergetics in ovarian cancer, «Molecular systems biology», 2014, 10, 5, p. 728; K. Yizhak, S.E. Le Dévédec, V.M. Rogkoti et al., A computational study of the Warburg effect identifies metabolic targets inhibiting cancer migration, «Molecular systems biology», 2014, 10, p. 744.